Implémentation en R du modèle
Gabriel Crépeault-Cauchon
2019-12-21
implementation.RmdImportation des données
Les données simulées sont disponibles à même le package. D’autres packages doivent aussi être importés.
library(xgbmr)
library(tidyverse)
#> ── Attaching packages ───────────────────────────────────────────────────── tidyverse 1.3.0 ──
#> ✔ ggplot2 3.2.1 ✔ purrr 0.3.3
#> ✔ tibble 2.1.3 ✔ dplyr 0.8.3
#> ✔ tidyr 1.0.0 ✔ stringr 1.4.0
#> ✔ readr 1.3.1 ✔ forcats 0.4.0
#> ── Conflicts ──────────────────────────────────────────────────────── tidyverse_conflicts() ──
#> ✖ dplyr::filter() masks stats::filter()
#> ✖ dplyr::lag() masks stats::lag()
library(xgboost)
#>
#> Attaching package: 'xgboost'
#> The following object is masked from 'package:dplyr':
#>
#> slice
library(mlr)
#> Loading required package: ParamHelpers
library(iml)
library(xtable) # pour des fins de présentation
library(gridExtra)
#>
#> Attaching package: 'gridExtra'
#> The following object is masked from 'package:dplyr':
#>
#> combine
dt <- SimulatedIndClaimsManipulation des données
Avant de pouvoir utiliser les données dans l’algorithme XGBoost, on doit d’abord effectuer un certain traitement des données.
Censuration des données
Dans la pratique, nous ne connaissons pas encore le vrai montant à l’ultime pour les réclamations qui sont plus récentes. Cet aspect a été tenu en compte, et une fonction du package permet de censurer les réclamations pour lesquelles nous ne sommes pas censé connaître l’information
set.seed(20191024) # Même seed que dans presentation-data.Rnw
dat %>%
select(ClNr, AY, starts_with('Pay'), real_ultimate) %>%
sample_n(5) %>%
knitr::kable()| ClNr | AY | Pay00 | Pay01 | Pay02 | Pay03 | Pay04 | Pay05 | Pay06 | Pay07 | Pay08 | Pay09 | Pay10 | Pay11 | real_ultimate |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 10031 | 1997 | 0 | 345 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | 345 |
| 26457 | 2002 | 1015 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | 1015 |
| 36641 | 2005 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 |
| 5903 | 1995 | 542 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | 542 |
| 15129 | 1999 | 200 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | 200 |
Modèle C : estimation des LDF
set.seed(20191023)
index <- makeResampleInstance(
desc = makeResampleDesc('Holdout', stratify.cols = 'AY', split = 0.70),
# il faut absolument donner une tâche et un target bidon
task = makeRegrTask(data = dt, target = 'Pay00')
)
traindt <- dat[index$train.inds[[1]],]
testdt <- dat[index$test.inds[[1]],]mack_bs_tri <- traindt %>%
getAggrTriangle() %>%
ChainLadder::BootChainLadder(R = 1000)
#>
#> Welcome to ChainLadder version 0.2.10
#>
#> Type vignette('ChainLadder', package='ChainLadder') to access
#> the overall package documentation.
#>
#> See demo(package='ChainLadder') for a list of demos.
#>
#> More information is available on the ChainLadder project web-site:
#> https://github.com/mages/ChainLadder
#>
#> To suppress this message use:
#> suppressPackageStartupMessages(library(ChainLadder))
ldf_bs <- mack_bs_tri$simClaims %>%
apply(3, function(triangle){
triangle %>% incr2cum() %>% as.triangle() %>% getLDF()
}) %>% t() %>% apply(2, quantile, probs = 0.8)
data.frame(LDF = ldf_bs) %>% t() %>% knitr::kable()| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| LDF | 2.0875 | 1.3137 | 1.1673 | 1.1081 | 1.07792 | 1.0533 | 1.0376 | 1.0252 | 1.0168 | 1.0121 | 1.0066 | 1 |
Préparer les jeux de données
train_a <- traindt %>%
widetolong() %>%
filter(complete.cases(.)) %>% # garder les années connues
filter(!(Open == 1 & Paid == 0)) %>% # enlever masse à 0
select(-AY, -TotalPaid, -Pay, -still, -max_dev_yr) %>%
rename(Ultimate = real_ultimate) %>%
# Arranger le dataset final pour le modèle A
select(ClNr:RepDel, devYear, Paid, Open, Ultimate) %>%
as_tibble()
train_b <- traindt %>%
widetolong() %>%
filter(complete.cases(.)) %>%
mutate(Ultimate = TotalPaid) %>%
filter(!(Open == 1 & Paid == 0)) %>% # enlever masse à 0
filter(Open == 0) %>%
select(-AY, -TotalPaid, -Pay, -still, -max_dev_yr, -real_ultimate) %>%
as_tibble()
train_c <- traindt %>%
widetolong() %>%
filter(complete.cases(.)) %>% # garder les années connues
mutate(Ultimate = case_when(
still == 1 ~ TotalPaid * ldf_bs[max_dev_yr + 1],
TRUE ~ TotalPaid)) %>%
filter(!(Open == 1 & Paid == 0)) %>% # enlever masse à 0
select(-AY, -TotalPaid, -Pay, -still, -max_dev_yr, -real_ultimate) %>%
as_tibble() # pour enlever le tag grouped dfIdem pour le test set :
Entrainement
Étant donné que les modèles xgboost peuvent être long à rouler, des modèles déjà entrainés sur les données d’exemple sont inclus avec le package. Les hyperparamètres des modèles sont disponibles dans la table suivante :
fit_a$learner$par.vals %>% data.frame(Modèle = 'A', .) %>%
bind_rows(fit_b$learner$par.vals %>% data.frame(Modèle = 'B', .),
fit_c$learner$par.vals %>% data.frame(Modèle = 'C', .)) %>%
knitr::kable()
#> Warning in bind_rows_(x, .id): Unequal factor levels: coercing to character
#> Warning in bind_rows_(x, .id): binding character and factor vector, coercing
#> into character vector
#> Warning in bind_rows_(x, .id): binding character and factor vector, coercing
#> into character vector
#> Warning in bind_rows_(x, .id): binding character and factor vector, coercing
#> into character vector| Modèle | nrounds | verbose | objective | eval_metric | eta | max_depth | min_child_weight | subsample | colsample_bytree |
|---|---|---|---|---|---|---|---|---|---|
| A | 1184 | 0 | reg:squarederror | rmse | 0.1 | 3 | 20 | 0.5 | 0.5 |
| B | 66 | 0 | reg:squarederror | rmse | 0.1 | 3 | 20 | 0.5 | 0.5 |
| C | 740 | 0 | reg:squarederror | rmse | 0.1 | 3 | 20 | 0.5 | 0.5 |
Note : la vignette entrainement_modeles permet d’avoir un peu plus de détail sur la procédure.
Résultats
test_dt <- test %>% select(-ClNr)
test_tsk <- makeRegrTask(data = test_dt, target = 'Ultimate')
#> Warning in makeTask(type = type, data = data, weights = weights, blocking =
#> blocking, : Provided data is not a pure data.frame but from class tbl_df, hence
#> it will be converted.Les modèles sont déjà inclus dans le package pour l’exemple
pred_a <- predict(fit_a, test_tsk)
pred_b <- predict(fit_b, test_tsk)
pred_c <- predict(fit_c, test_tsk)Mettre ensemble les variables explicatives et les prédictions
test_pred_a <- test_dt %>%
rownames_to_column('id') %>%
mutate_at(vars(id), as.integer) %>%
left_join(pred_a$data, by = "id")
test_pred_b <- test_dt %>%
rownames_to_column('id') %>%
mutate_at(vars(id), as.integer) %>%
left_join(pred_b$data, by = "id")
test_pred_c <- test_dt %>%
rownames_to_column('id') %>%
mutate_at(vars(id), as.integer) %>%
left_join(pred_c$data, by = "id")Distribution prédictive des réserves totales (Bootstrap)
## Vraie réserve (selon données simulées du test set)
vraie_reserve <- test_dt %>% summarise(paye = sum(Paid), total = sum(Ultimate), R = total - paye)
## Calcul des réserves totales selon chaque modèle
aa <- apply(bs_pred_a, 2, sum)
bs_res_a <- (aa - sum(test_dt$Paid)) %>% data.frame(A = .)
bb <- apply(bs_pred_b, 2, sum)
bs_res_b <- (bb - sum(test_dt$Paid)) %>% data.frame(B = .)
cc <- apply(bs_pred_c, 2, sum)
bs_res_c <- (cc - sum(test_dt$Paid)) %>% data.frame(C = .)Et on obtient le tableau suivant :
bind_cols(bs_res_a, bs_res_b, bs_res_c) %>%
gather('Modèle', 'Réserve') %>%
group_by(Modèle) %>%
nest() %>%
mutate(
moyenne = map_dbl(data,function(x) unlist(x) %>% mean),
sd = map_dbl(data,function(x) unlist(x) %>% sd),
VaR95 = map_dbl(data,function(x) unlist(x) %>% quantile(., probs = .95)),
VaR99 = map_dbl(data,function(x) unlist(x) %>% quantile(., probs = .99))
) %>%
select(-data) %>%
data.frame() %>%
format(big.mark = ' ', justify = 'centre') %>%
knitr::kable()| Modèle | moyenne | sd | VaR95 | VaR99 |
|---|---|---|---|---|
| A | 25 482 234 | 1 720 920 | 27 927 106 | 27 951 939 |
| B | 2 937 217 | 1 653 261 | 5 326 843 | 5 885 246 |
| C | 24 057 011 | 1 098 914 | 25 779 615 | 26 007 870 |
Et le graphique suivant :
bind_cols(bs_res_a, bs_res_b, bs_res_c) %>%
ggplot() +
geom_density(aes(x = A, y = ..density.., fill = 'A'), n = 2^12, alpha = 0.4) +
geom_density(aes(x = B, y = ..density.., fill = 'B'), n = 2^12, alpha = 0.4) +
geom_density(aes(x = C, y = ..density.., fill = 'C'), n = 2^12, alpha = 0.4) +
theme_classic() +
theme(legend.position = 'bottom') +
labs(fill = 'Modèles', x = 'Réserve totale', y = 'Probabilité') +
geom_vline(xintercept = vraie_reserve$R, linetype = 'dashed')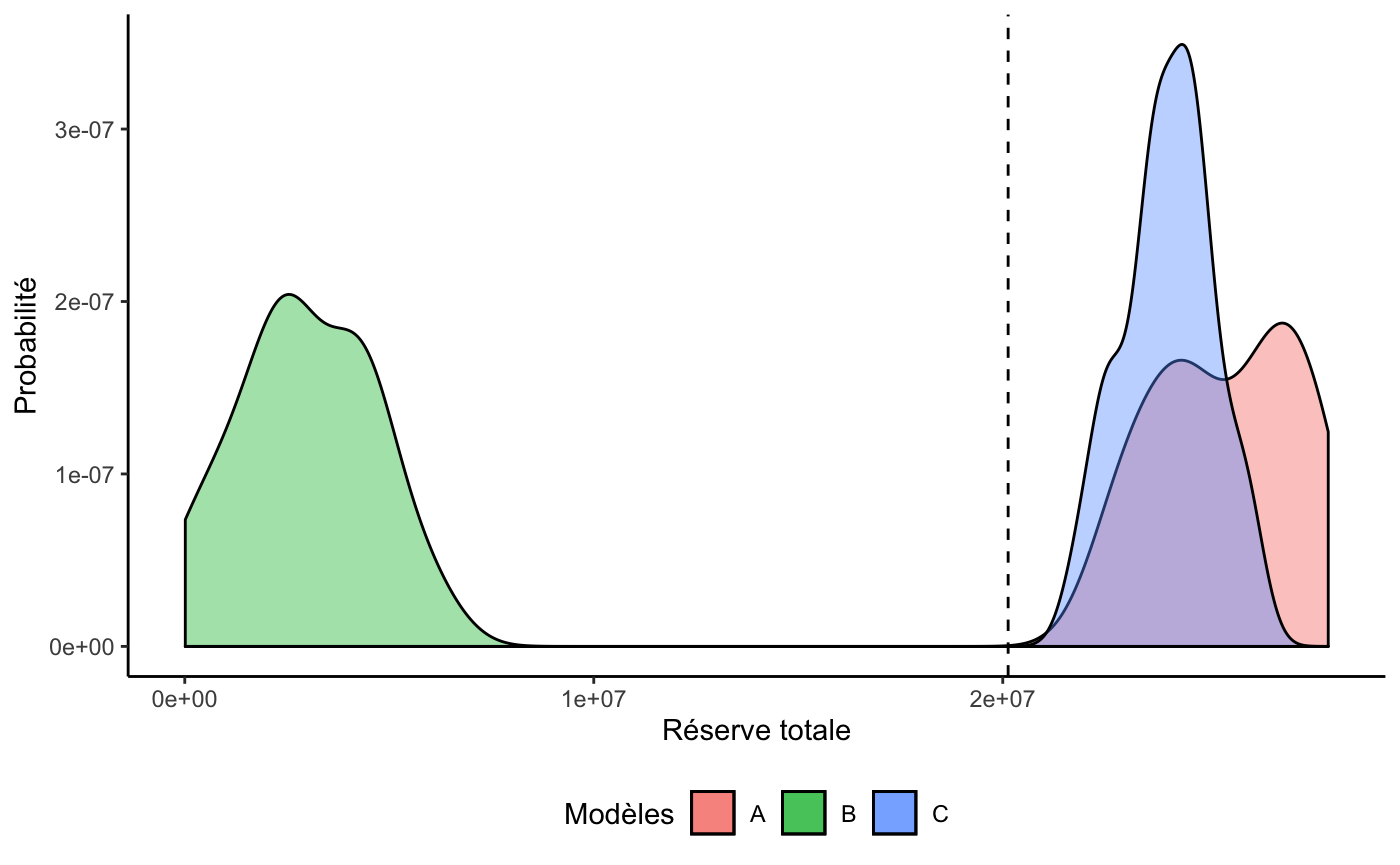
Graphique des prédictions en fonction de la vraie valeur
zoom_out_pred <- pred_c$data %>%
# filter(response > 0) %>%
ggplot(aes(x = truth, y = response)) +
geom_point() +
annotate('rect',
xmin = 0, xmax = 200000,
ymin = 0, ymax = 200000,
alpha = 0.3, col = 'blue') +
# geom_vline(xintercept = qq, col = 'red', linetype = 'dashed') +
geom_abline(intercept = 0, slope = 1,
col = 'blue', size = 1, linetype = 'dashed') +
# coord_cartesian(xlim = c(0, 25000), ylim = c(0,25000)) +
labs(y = 'Valeur prédite')
zoom_in_pred <- pred_c$data %>%
# filter(response > 0) %>%
ggplot(aes(x = truth, y = response)) +
geom_point() +
# geom_vline(xintercept = qq, col = 'red', linetype = 'dashed') +
geom_abline(intercept = 0, slope = 1,
col = 'blue', size = 1, linetype = 'dashed') +
coord_cartesian(xlim = c(0, 200000), ylim = c(0,200000)) +
labs(y = 'Valeur prédite')
zoom <- arrangeGrob(zoom_out_pred, zoom_in_pred)
plot(zoom)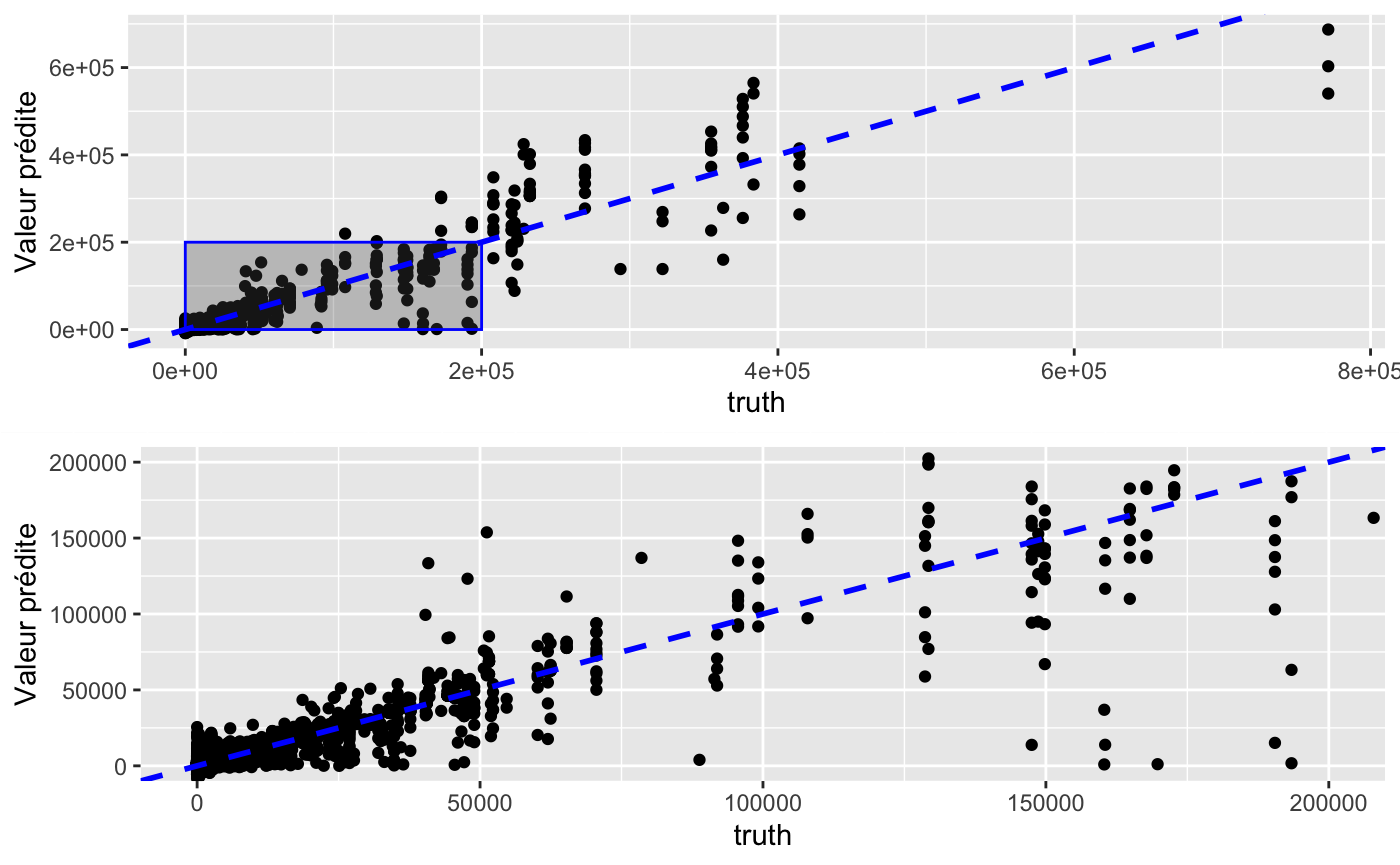
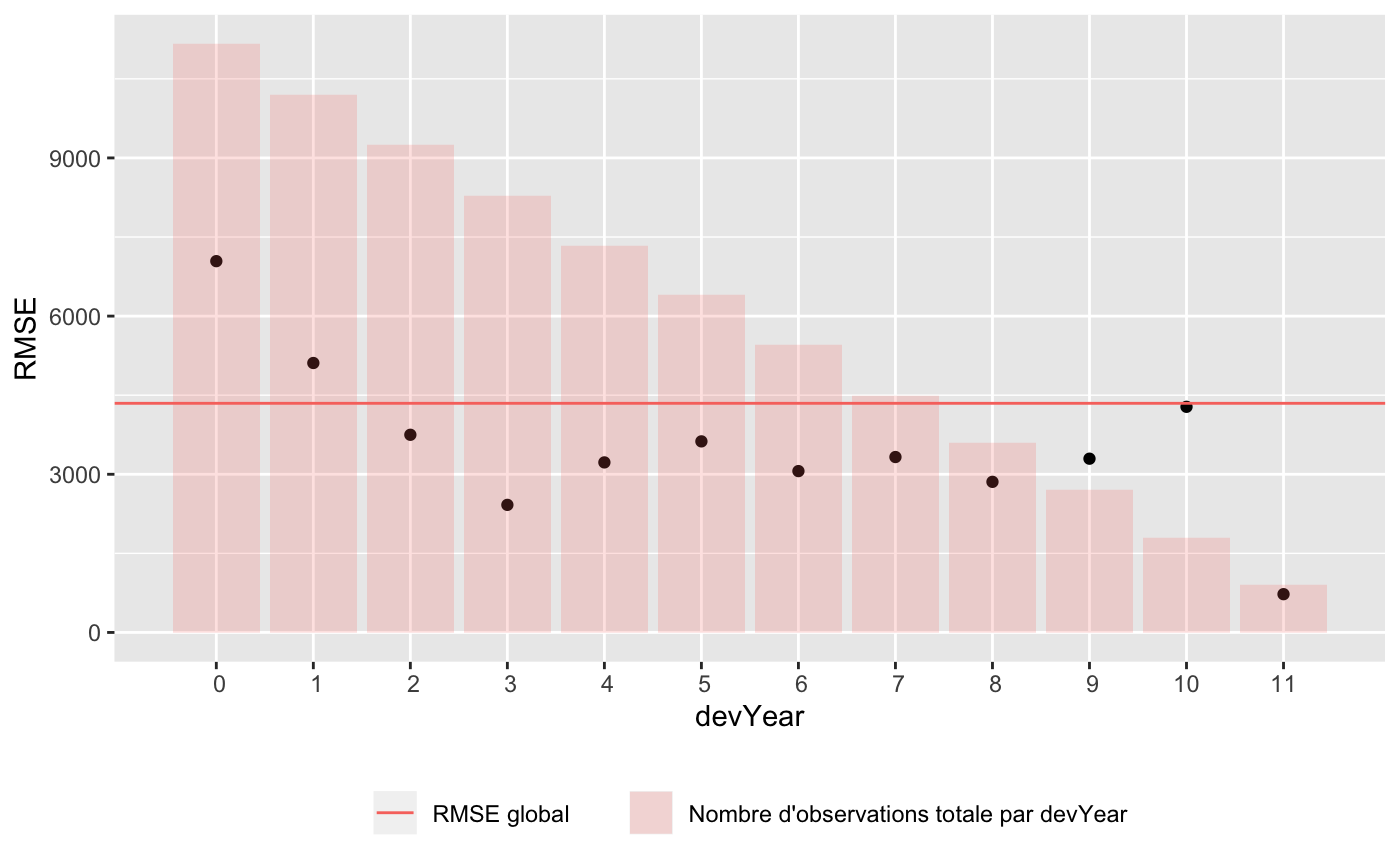
Prediction Lift
pred_a$data %>%
rename(A = response) %>%
mutate(
B = pred_b$data$response,
C = pred_c$data$response
) %>%
gather('Modèle', 'response', A,B,C) %>%
group_by(Modèle) %>% nest() %>%
mutate(quantile = map(data, function(dt){
dt %>%
transmute(quantile = ntile(response, n = 5))
})) %>%
unnest(cols = c(data, quantile)) %>% select(-id) %>%
group_by(Modèle, quantile) %>%
summarise(
reel_moyen = mean(truth),
rmse_moyen = sqrt(mean((truth - response)^2))
) %>%
mutate(quantile = case_when(
quantile == 1 ~ "(0, 20)",
quantile == 2 ~ "(20, 40)",
quantile == 3 ~ "(40, 60)",
quantile == 4 ~ "(60, 80)",
quantile == 5 ~ "(80, 100)"
)) %>%
ggplot(aes(x = quantile)) +
geom_bar(aes(y = reel_moyen),stat = 'identity', fill = 'green',
alpha = 0.4, col = 'black') +
geom_line(aes(y = rmse_moyen, col = 'RMSE moyen par quantile'),
linetype = 'dashed', group =1) +
geom_point(aes(y = rmse_moyen), col = 'red') +
theme(legend.position = 'bottom',
axis.text = element_text(angle = 90, hjust = 1, vjust = 0.5)) +
coord_cartesian(ylim = c(0, 15000)) +
labs(
y = "Valeur réelle moyenne",
x = "Regroupement des quantiles de la variable réponse", col = NULL) +
facet_grid(. ~ Modèle)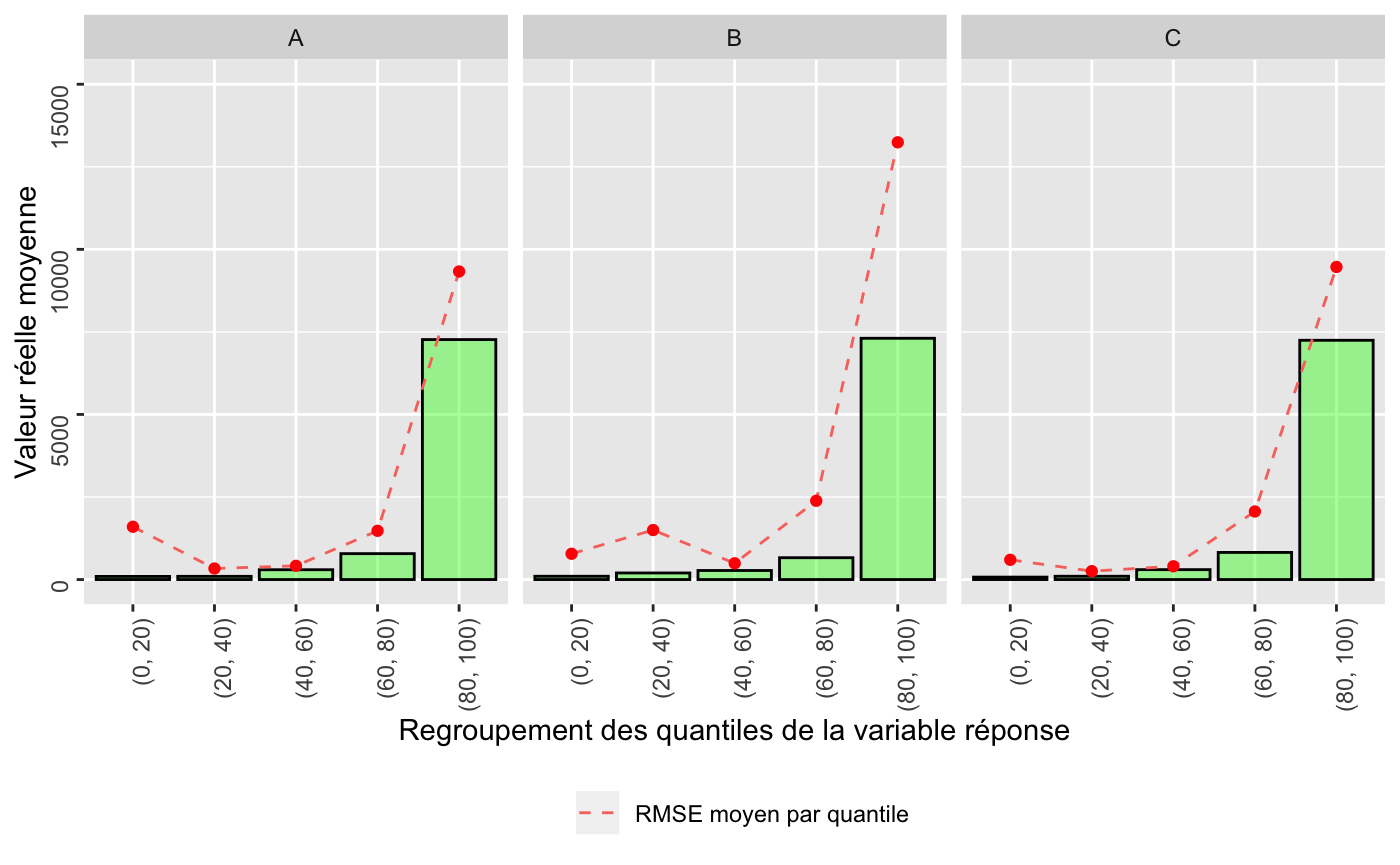
Graphique des résidus du modèle C
set.seed(2019)
pred_a$data %>%
sample_frac(.1) %>%
mutate(erreur = truth - response) %>%
ggplot() +
geom_histogram(aes(x = erreur), binwidth = 100) +
coord_cartesian(xlim = c(-2000, 2000)) +
labs(x = expression(epsilon = y[i] - hat(y)[i]),
y = 'Fréquence',
caption = 'Échantillon aléatoire de 10% des prédictions sur les données de validation') +
scale_y_discrete(limits = seq(0, 2000, 100)) 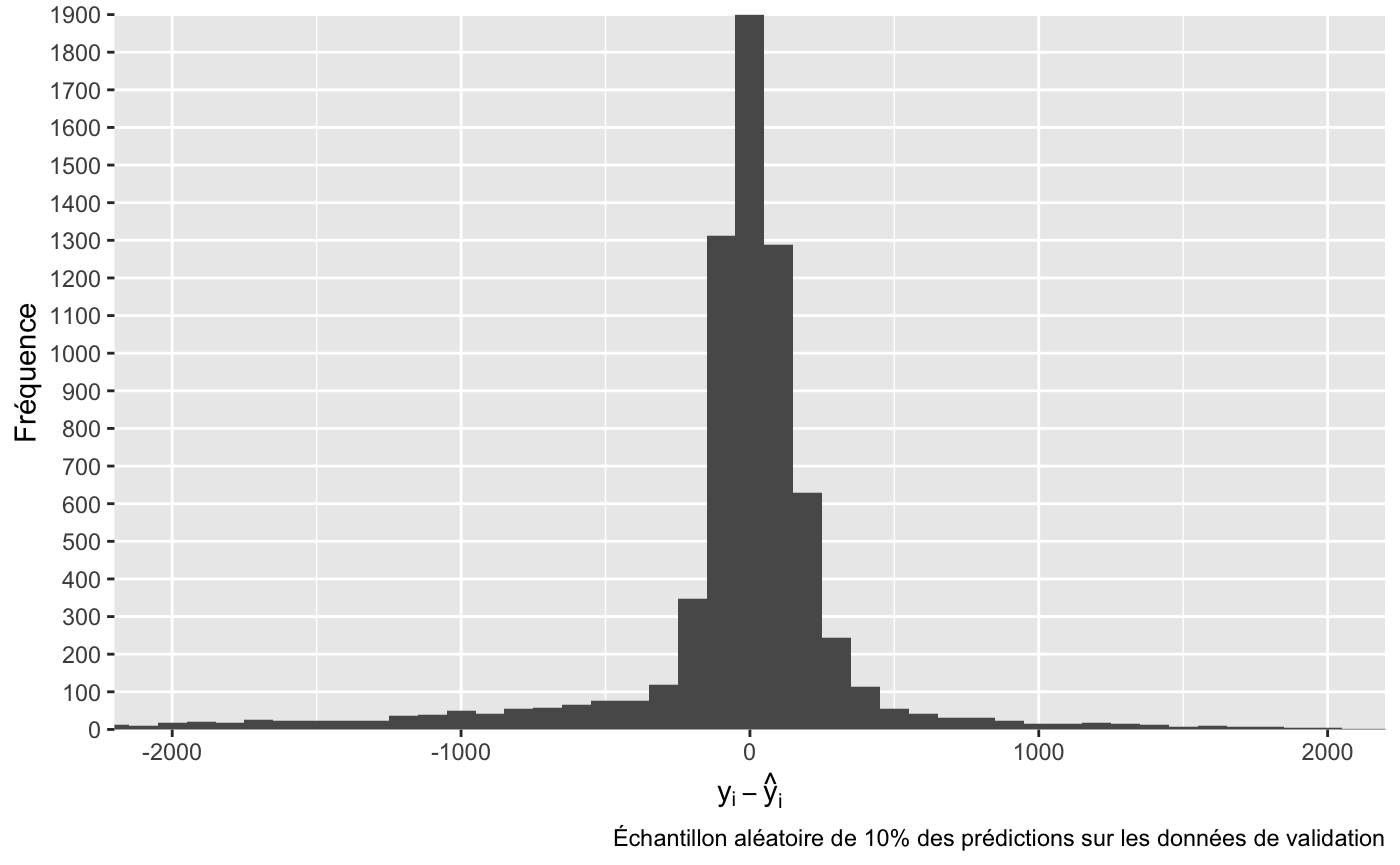
Interptétabilité des modèles
set.seed(2019)
predictor_a <- test_dt %>%
# On sélectionne 30 observations par année de développement
group_by(devYear) %>% sample_n(30) %>%
# on rammène en tibble car iml n'accepte pas un 'grouped_df' de dplyr
as_tibble() %>%
Predictor$new(model = fit_a, data = select(., -Ultimate), y= select(., Ultimate))
predictor_b <- test_dt %>%
# On sélectionne 30 observations par année de développement
group_by(devYear) %>% sample_n(30) %>%
# on rammène en tibble car iml n'accepte pas un 'grouped_df' de dplyr
as_tibble() %>%
Predictor$new(model = fit_b, data = select(., -Ultimate), y= select(., Ultimate))
predictor_c <- test_dt %>%
# On sélectionne 30 observations par année de développement
group_by(devYear) %>% sample_n(30) %>%
# on rammène en tibble car iml n'accepte pas un 'grouped_df' de dplyr
as_tibble() %>%
Predictor$new(model = fit_a, data = select(., -Ultimate), y= select(., Ultimate))Graphique des Feature importance
varimp_a <- FeatureImp$new(predictor_a, loss = "mse") %>% plot() + theme_classic()
varimp_b <- FeatureImp$new(predictor_b, loss = "mse") %>% plot() + theme_classic()
varimp_c <- FeatureImp$new(predictor_c, loss = "mse") %>% plot() + theme_classic()
# grid.arrange(varimp_a, varimp_b, varimp_c, nrow = 3)
varimp_c
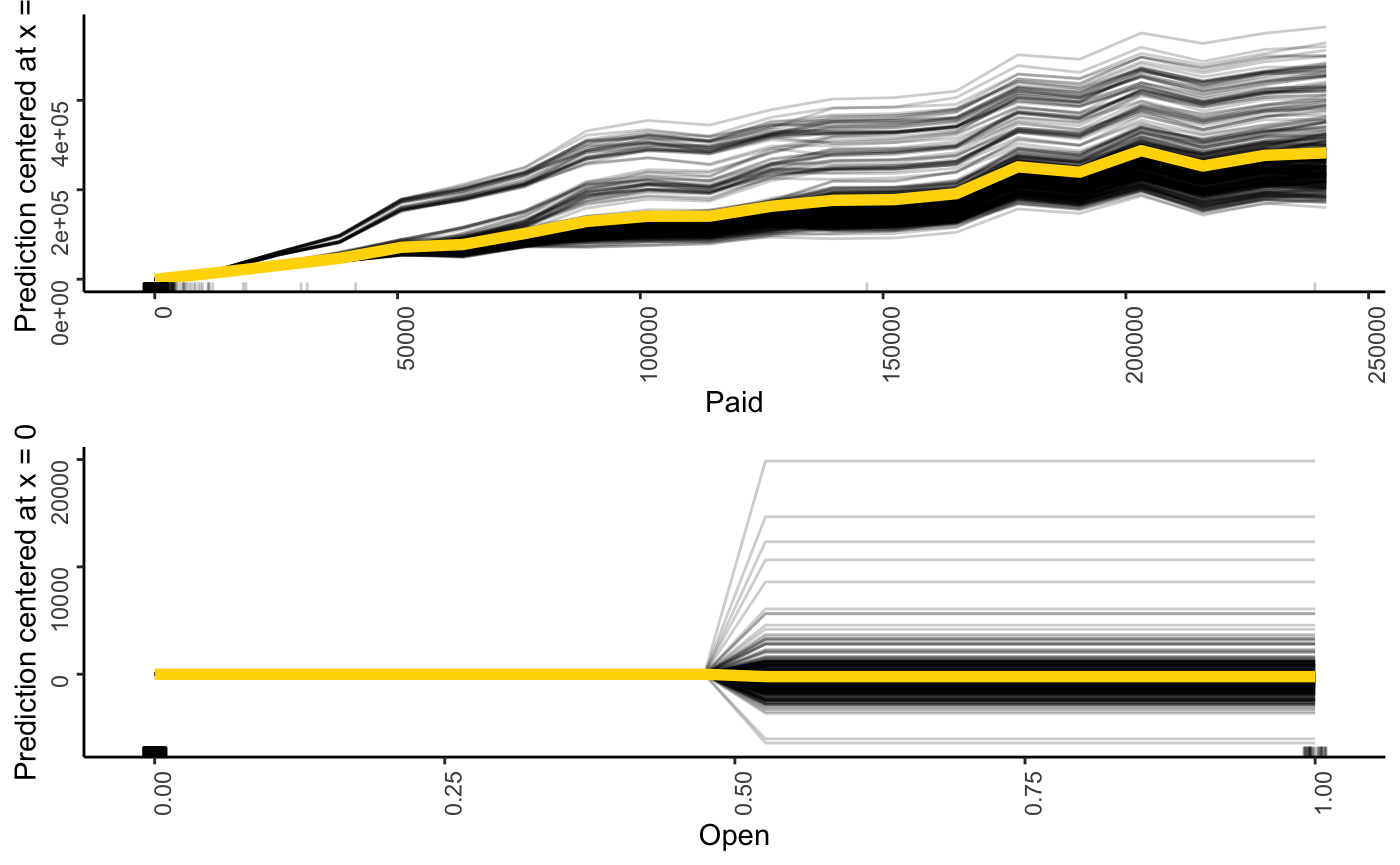
PDP/ICE plots
var2check <- c('Paid', 'Open')
pdp_ice <- lapply(var2check,
function(var){
FeatureEffect$new(predictor_a,
feature = var,
method = 'pdp+ice',
center.at = 0) %>%
plot() + theme_classic() +
theme(axis.text = element_text(angle = 90, hjust = 1, vjust = 0.5))
})
# Produire les 4 graphiques dans une seule figure.
do.call('grid.arrange', pdp_ice)